Las proteínas están formadas por cadenas de aminoácidos, y en cada proteína se pueden distinguir secuencias de aminoácidos cortas con una función discreta, llamadas motivos funcionales. En ocasiones estos motivos ya están descritos, y en otras están por definir. Cuando un motivo funcional aparece modificado, es posible que esa mutación influya en el desarrollo de una enfermedad como el cáncer. Verificar los posibles cambios en una proteína es uno de los primeros pasos parar llevar adelante una investigación sobre su función. Si tenemos en cuenta que el actual borrador del proteoma humano está formado por más de 40.000 proteínas, buscar las mutaciones en cada una supone una tarea ingente.
Por esta razón, los genetistas Asier Fullaondo y José Antonio Rodríguez, junto al ingeniero de Telecomunicaciones Gorka Prieto de la Universidad del País Vasco (UPV/EHU), han creado WREGEX 2.0, una aplicación bioinformática, versátil y fácil de usar, para analizar y combinar la información de 40.000 proteínas en un minuto. Está disponible para la comunidad científica en el servidor de la universidad y la revista Scientific Reports ha publicado un artículo sobre ella.
La herramienta se denomina WREGEX 2.0 y es capaz de analizar 40.000 proteínas en un minuto
Los tres investigadores comenzaron sumando sus fuerzas: Rodríguez tenía la pregunta biológica, Fullaondo el conocimiento de herramientas y bases de datos bioinformáticas, y Prieto la capacidad de programación. En un principio, los doctores desarrollaron un software (WREGEX) que sirve para predecir y buscar de manera automática ‘motivos funcionales’ (los pequeños grupos de aminoácidos que desarrollan tareas específicas de una proteína).
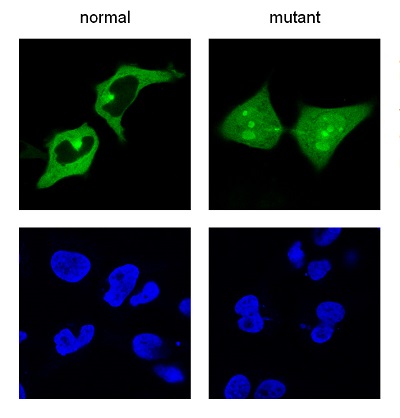
Probaron el programa para predecir ‘motivos’ que mueven una proteína desde el núcleo al citoplasma de una célula, las llamadas ‘señales de exportación nuclear’. Al finalizar esta fase de investigación en 2014, publicaron un artículo en la revista Bioinformatics. Pero, como dice José Antonio Rodríguez, “en investigación la respuesta a una pregunta abre la puerta a más preguntas”. La pregunta en esta ocasión fue: ¿Qué proteínas pueden tener en su secuencia de aminoácidos un ‘motivo’ funcional mutado en cáncer?
El equipo dio un paso más y combinó la información sobre la secuencia de todas las proteínas humanas conocidas con el catálogo COSMIC que recoge las mutaciones relacionadas con el cáncer. Así, aparece una nueva versión (WREGEX 2.0) que permite comparar una proteína normal y la misma mutada para, de esta forma, poder predecir ‘motivos funcionales’ que hayan sido modificados y puedan tener una relación con el cáncer. “También puedes tener experiencia en el funcionamiento de un motivo y quieres conocer en qué proteínas puede aparecer y si aparece modificado en cáncer. Mediante este software obtienes candidatos para empezar a estudiar”, explica Gorka Prieto.
Ensayo de exportación en células
Una vez desarrollado el programa bioinformático había que probarlo, y, para ello, realizaron un ‘ensayo de exportación en células´. Volvieron a escoger varios candidatos que pudieran constituir un motivo encargado de mover la proteína fuera del núcleo de la célula. Comprobaron su funcionamiento y, tras modificarlos según las mutaciones tumorales descritas en el catálogo COSMIC, volvieron a realizar el ensayo. De esta manera, certificaron que los candidatos actuaban como una ‘señal de exportación’, que la mutación afectaba a su funcionamiento; y, en consecuencia, que el software era válido. La herramienta, por tanto, combina tres tipos de informaciones: las secuencias de proteínas, los motivos funcionales y las mutaciones del cáncer.
“Una de las principales características de WREGEX 2.0 es que puede estudiar simultáneamente proteomas muy complejos, con muchísimas proteínas, y combinar información, en el caso del ensayo, con mutaciones de cáncer; pero la puerta está abierta a utilizar otras bases de datos que contengan información sobre otro tipo de mutaciones. La ventaja, además, es que en un minuto se pueden analizar 40.000 proteínas, mientras que con otros programas el análisis de una proteína llevaba varios minutos”, explica Asier Fullaondo. Por tanto, mediante este software, se puede predecir que la alteración en una proteína puede influir en el desarrollo de la enfermedad, no sólo cáncer.
Hasta ahora, ya hay trece trabajos de investigación que han utilizado esta herramienta informática. Y al servidor han entrado investigadores de China, Japón, Corea, Alemania y Estados Unidos. Mientras tanto, el tándem multidisciplinar formado por los tres doctores ya piensa en seguir trabajando para mejorar la herramienta.
Referencia bibliográfica
Gorka Prieto, Asier Fullaondo & Jose A. Rodríguez. «Proteome-wide search for functional motifs altered in tumors: Prediction of nuclear export signals inactivated by cancer-related mutations». Scientific Reports, 2016. doi:10.1038/srep25869