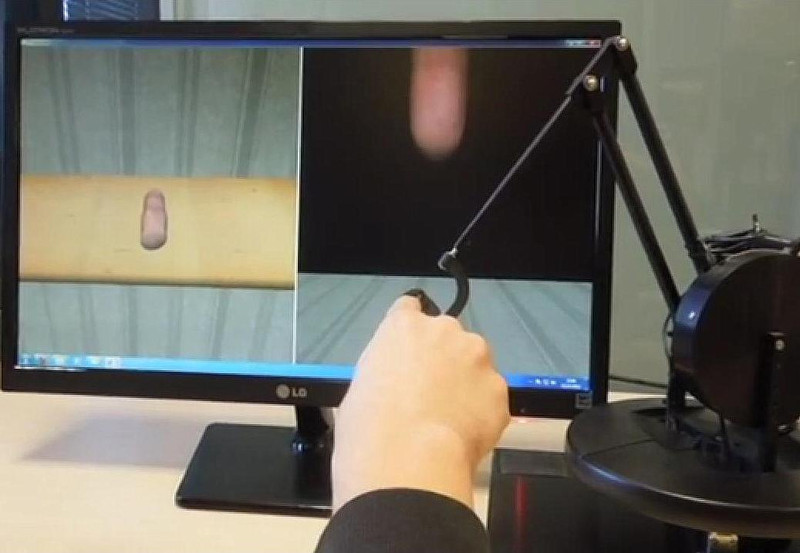
La simulación del movimiento de un líquido, una tela, un órgano o un sistema biológico está regida por leyes mecánicas similares a las de los videojuegos y requiere la resolución de problemas geométricos parecidos. En este marco, investigadores del Multimodal Simulation Lab de la Universidad Rey Juan Carlos (URJC), capitaneados por el profesor Miguel Ángel Otaduy, simulan deformaciones, fluidos, fracturas o colisiones.
La simulación de estos fenómenos tiene aplicaciones diversas en el mundo de los gráficos por ordenador, la ingeniería y la medicina; en concreto en la animación, los videojuegos, el prototipado virtual y el entrenamiento médico. Algunos avances sobre los métodos de simulación los han publicado en la revista Computer Graphics Forum y detalles sobre como visualizar los datos en IEEE Transactions on Visualization and Computer Graphics. También presentan sus resultados en un canal de Youtube.
“El conocimiento fundamental en esos aspectos es trasladable de un problema a otro y nos permite acometer aplicaciones de la simulación aparentemente muy dispares. Por otro lado, el conocimiento aplicado reside en los expertos clínicos, biólogos o artistas en cada caso y el desarrollo de soluciones de éxito requiere obviamente la colaboración de expertos”, explica el profesor Otaduy.
Este tipo de fenómenos han sido estudiados durante siglos, pero el hilo principal y novedoso de su investigación, desde el punto de vista de los gráficos por computador, es que las simulaciones producen un contenido interactivo. Los casos más conocidos, en aplicaciones de entretenimiento, son la animación por ordenador y los videojuegos. Pero existen más ejemplos donde se pueden aplicar estos procesos, como los juegos para aprendizaje, el entrenamiento y la planificación en medicina, el diseño computacional basado en simulación o simulación biomecánica para tacto virtual.
Buscar nuevos modelos y algoritmos
En la actualidad, para desarrollar este tipo de modelos se necesitan supercomputadoras, los procesos son complejos y pueden tardar meses e incluso años. Es por ello que uno de los principales retos planteados por los investigadores consiste en buscar nuevos modelos y algoritmos computacionalmente más eficientes.
“A veces el objetivo es producir efectos visuales que antes no eran posibles salvo bajo computadoras carísimas y procesos lentísimos, otras veces el objetivo es alcanzar simulaciones interactivas minimizando la pérdida de calidad, y un objetivo particular en los gráficos por computador es obtener simulaciones controlables, que se comporten como el usuario desee dentro de lo físicamente plausible”, según indica el profesor Otaduy.
El proyecto europeo Animetrics que dirige, financiado por el programa IDEAS de las prestigiosas ERC Starting Grant, busca simplificar los modelos existentes, utilizando datos precalculados o medidos de la realidad para desarrollar modelos y algoritmos de simulación más potentes. En otro proyecto, denominado Wearhap, desarrollan modelos interactivos de la biomecánica de la mano humana para aplicaciones en tacto virtual.
“También trabajamos en simulación de telas extremadamente realistas para distintas aplicaciones, simulación anatómica para planificación clínica sobre imagen médica, dinámica de fluidos para animación por ordenador o incluso aceleración de algoritmos de dinámica molecular” explica el investigador, quien añade que “como los problemas a los que nos enfrentamos normalmente tienen un componente visual importante, un factor bastante común en nuestras soluciones es adaptar la descripción mecánica de manera inteligente a la geometría del problema”.
Por otro lado, han trabajado con empresas dedicadas al desarrollo de simulaciones de sistemas biológico, como la empresa Plebiotic, para adaptar métodos tradicionales de simulación de dinámica molecular con el objetivo de poder ejecutarlos de manera masivamente paralela sobre tarjetas gráficas. La colaboración con la empresa Plebiotic ha dado lugar a un programa de diseño de fármacos llamado PleMD. Actualmente su trabajo consiste en paralelizar la computación sobre múltiples tarjetas gráficas para ser capaces de acometer la simulación de sistemas biológicos con millones de átomos.
“La dinámica molecular sucede a escalas espaciales y temporales radicalmente distintas de otros fenómenos mecánicos que estudiamos. Por poner un ejemplo, el movimiento de los átomos de una proteína se ha de resolver a una resolución espacial del orden del angstrom (hay 10 millones de angstrom en 1 milímetro) y a una resolución temporal de 1 femtosegundo (hay mil billones de femtosegundos en 1 segundo)”, expone el profesor de la URJC.
“Para tratar de predecir el comportamiento de una proteína y/o un virus viendo su movimiento era necesario ejecutar simulaciones de meses de duración en supercomputadoras. En los últimos años, el desarrollo de las tarjetas gráficas como plataformas de computación masivamente paralela ha hecho posible que estas simulaciones se puedan ejecutar en ordenadores cotidianos. Pero esto no sucede por sí solo”, añade.
(URJC)
Referencias bibliográficas:
‘A Survey on Position-Based Simulation Methods in Computer Graphics’, Jan Bender, Matthias Müller, Miguel A. Otaduy, Matthias Teschner, Miles Macklin. Computer Graphics Forum – 2014
‘Axis Calibration for Improving Data Attribute Estimation in Star Coordinates Plots’, Manuel Rubio-Sánchez, Alberto Sánchez, IEEE Transactions on Visualization and Computer Graphics (Proceedings of Scientific Visualization / Information Visualization 2014), Volume 20, Number 12 – dec 2014