Científicos del Grupo de Cristalografía de Macromoléculas del Centro Nacional de Investigaciones Oncológicas (CNIO), dirigido por Guillermo Montoya, ha desarrollado un método de producción de cristales biológicos que ha permitido observar, por primera vez, cómo se produce la rotura de la doble cadena del ADN.
Además, han desarrollado una simulación informática que hace visible al ojo humano este proceso, de duración en la naturaleza de millonésimas de segundo. El trabajo se publica hoy en la revista Nature Structural & Molecular Biology.
“Se sabía que las enzimas, o proteínas, endonucleasas son las responsables de esta rotura, pero hasta ahora no se conocía el mecanismo exacto”, cuenta Montoya. “En nuestro trabajo detallamos los procesos dinámicos de esta reacción biológica básica mediada por la enzima I-DmoI. Nuestras observaciones son extrapolables a otras muchas familias de endonucleasas que actúan de forma idéntica”.
La rotura del ADN ocurre en numerosos procesos naturales clave para la vida: mutagénicos, de síntesis, de recombinación, de reparación. El campo de la biología molecular la emplea, también, en procesos artificiales.
Una vez desentrañado el mecanismo exacto por el que se produce, este conocimiento podrá emplearse en múltiples aplicaciones biotecnológicas: desde la corrección de mutaciones para tratar enfermedades raras y genéticas, hasta el desarrollo de organismos genéticamente modificados.
Reacción en slow motion
Las enzimas son sistemas dinámicos altamente especializados. Su función de rotura podría asemejarse, según indica Montoya, a la de una máquina para cortar telas tan especializada, que estuviera programada “para hacer el corte solo cuando bajo la cuchilla pasara un trozo de tejido con una determinada combinación de colores”.
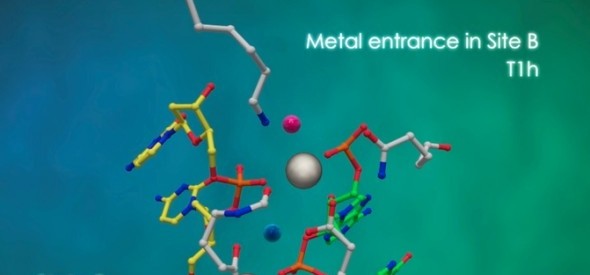
En este caso, los investigadores se centraron en observar los cambios conformacionales ocurridos en el centro activo de I-DmoI, aquella región que contiene los aminoácidos que actúan como cuchilla e inducen la rotura del ADN.
Alterando la temperatura y el pH, el equipo del CNIO ha ralentizado hasta diez días una reacción química que en condiciones normales dura microsegundos. En estas condiciones, han creado una película en slow-motion de todo el proceso.
“Introduciendo metales hemos sido capaces de disparar la reacción enzimática para posteriormente producir cristales biológicos y congelarlos a -200 ºC”, explica Montoya. “Así, recogimos hasta 185 estructuras cristalográficas que representan todos los cambios conformacionales de cada paso de la reacción”.
Finalmente, mediante análisis computacional, los investigadores han ilustrado los siete estadios intermedios del proceso de separación de las cadenas de ADN. “Es muy emocionante, porque la elucidación de este mecanismo nos dará la información necesaria para rediseñar estas enzimas y mejorar la precisión de los bisturíes moleculares, las herramientas esenciales para la modificación del genoma”, concluye.
El estudio ha contado con la colaboración del grupo computacional de Modesto Orozco, de IRB Barcelona, y ha sido financiado por el Ministerio de Economía y Competitividad y la Fundación Ramón Areces.
Referencia bibliográfica:
Visualizing phosphodiester-bond hydrolysis by an endonuclease. Rafael Molina, Stefano Stella, Pilar Redondo, Hansel Gomez, María José Marcaida, Modesto Orozco, Jesús Prieto & Guillermo Montoya. Nature Structural & Molecular Biology (2014). doi: 10.1038/nsmb.2932