Karina Toledo/Agência FAPESP Las cinasas o quinasas son enzimas que funcionan haciendo las veces de “interruptores” de las proteínas, y de esa forma regulan procesos importantes en el organismo, entre ellos la división, la proliferación y la diferenciación celular.
Cualquier problema en esa señalización puede resultar en patologías tales como el cáncer, inflamaciones o enfermedades cardiovasculares. Por este motivo, aproximadamente una cuarta parte de los esfuerzos de la industria farmacéutica apunta a desarrollar compuestos capaces de modular la actividad de esas enzimas.
Un estudio llevado a cabo por científicos brasileños, y destacado en la portada de la revista Science Signaling, puede llevar a que torne más fácil detectar a la proteína blanco (o sustrato) de una determinada cinasa, una tarea para nada trivial si se considera que en el genoma humano existen más de 500 enzimas distintas de este tipo.
“Cuando logramos identificar al sustrato de una cinasa podemos no sólo comprender mejor su función en el organismo, sino también diseñar compuestos capaces de modular su acción de manera más selectiva para causar menos efectos adversos”, afirmó Deborah Schechtman, docente del Instituto de Química de la Universidad de São Paulo (USP) y coordinadora de la investigación que contó con el apoyo de la FAPESP.
Las cinasas, explicó Schechtman, son las encargadas de una reacción química conocida como fosforilación, que consiste en transferir un grupo fosfato de moléculas de alta energía como el ATP (trifosfato de adenosina), a la proteína blanco. Esta reacción puede llevar –dependiendo del caso– a que se active la proteína, o a que se desactive, o incluso puede señalizar que la molécula debe ser degradada.
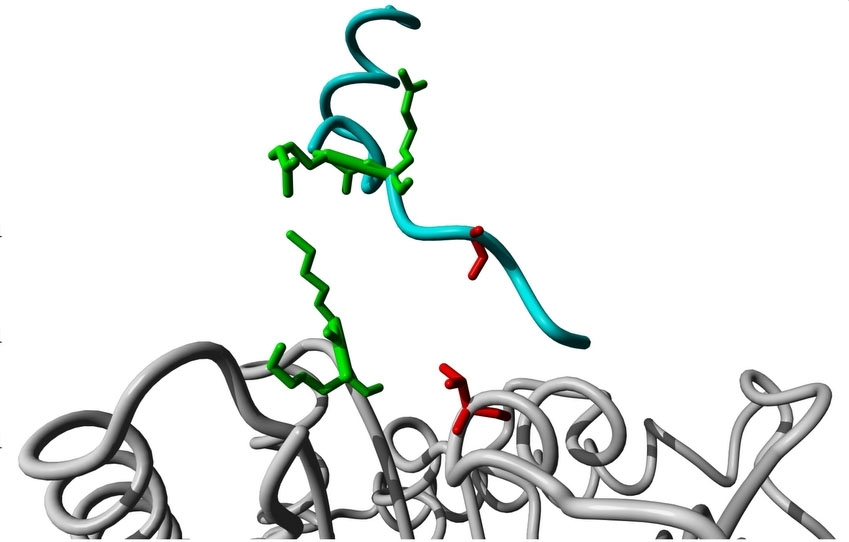
Con todo, para que se concrete esta interacción, la enzima y el sustrato deben encastrarse como si fuesen piezas de un rompecabezas. La proteína blanco debe tener una secuencia específica de aminoácidos –denominada técnicamente “secuencia consenso”– a la cual la cinasa pueda reconocerla.
“En las investigaciones anteriores se buscó esa secuencia consenso en la estructura lineal de la proteína [en la cadena de aminoácidos unidos químicamente entre sí]. Nuestro trabajo demostró que en algunos casos sólo es posible hallarla cuando se observa la estructura tridimensional de la molécula. Es un cambio de paradigma”, afirmó Schechtman.
Para que la proteína pueda desempeñar su función dentro de la célula, la cadena de aminoácidos que la compone debe plegarse y asumir una forma tridimensional específica, en un proceso conocido como ovillado.
Cuando esto ocurre, los aminoácidos que se encontraban alejados unos de otros en la estructura primaria pasan a acercarse y –en algunos casos– pueden formar aquello a lo que el grupo de Schechtman denominó consenso estructural, que también puede ser reconocido por la cinasa.
“Nosotros confirmamos que los llamados residuos anclas –que forman el consenso que será reconocido por la cinasa– son importantes para que se concrete la interacción entre la enzima y el sustrato, y pueden erigirse en objetos del desarrollo de drogas inhibidoras con actividad altamente específica”, afirmó Schechtman.
De acuerdo con la investigadora, los inhibidores actualmente disponibles en el arsenal farmacéutico impiden la transferencia del grupo fosfato a las proteínas, pero afectan a las cinasas del organismo de manera indiscriminada, por lo que pueden verse perjudicados ciertos procesos fisiológicos.
La tubulina
La línea de investigación que llevó al descubrimiento publicado en Science Signaling tuvo inicio en un proyecto anterior, también apoyado por la FAPESP, en el cual Schechtman procuraba descubrir sustratos de una cinasa conocida como PKC (proteína cinasa C), implicada en la proliferación y en la diferenciación de células madre embrionarias.
“Descubrí que la PKC interactuaba con una proteína llamada tubulina, que participa en el proceso de división celular. Entonces nos abocamos a investigar ese sustrato. Mediante el empleo de una técnica conocida como espectrometría de masa, detectamos con qué residuo de aminoácido de la tubulina interactuaba la PKC y nos percatamos de que no había a su alrededor una secuencia consenso de reconocimiento de la cinasa”, comentó la investigadora.
En colaboración con el grupo del investigador Paulo Sergio Lopes de Oliveira, del Laboratorio Nacional de Biociencias (LNBio), Schechtman decidió investigar la estructura tridimensional de la tubulina y encontró aquello a lo que denominó consenso estructural.
“Para confirmarlo, elaboramos algunas versiones mutadas de la tubulina, con residuos anclas distintos, y observamos que el reconocimiento de la cinasa se veía bastante perjudicado”, comentó Schechtman.
El paso siguiente consistió en probar que podrían hallar el consenso estructural también en otras proteínas y, para ello, los investigadores realizaron un trabajo de modelado.
“Como se trataba de probar un concepto y existen pocos sustratos conocidos para la PKC, realizamos ese trabajo de modelado con la PKA (proteína cinasa A), otra cinasa mejor estudiada. De ese modo, nuestra muestra sería mayor”, comentó Oliveira.
Los científicos analizaron entonces un banco de datos de sitios de fosforilación de la PKA detectados experimentalmente, hicieron comparaciones con información de un banco de estructuras tridimensionales de proteínas determinadas mediante cristalografía y recurrieron al modelado para ver de qué manera quedarían las secuencias en el espacio en tres dimensiones.
“Modelamos más de mil sustratos de PKA y observamos que en 27 casos los residuos anclas exhibían tan sólo el consenso estructural”, comentó Oliveira.
Los resultados del modelado también sugieren que, para que la proteína sea reconocida por la cinasa, no sólo debe estar correcto el orden de los aminoácidos en la estructura, sino que también la distancia entre ellos constituye un factor determinante. Es como si fuesen piezas de un rompecabezas.
Actualmente, el grupo de Schechtman sigue investigando la proteína tubulina en un intento por entender cuál es el efecto de la fosforilación que se concreta durante la división celular. El objetivo de la investigadora consiste en comprender mejor los procesos moleculares de diferenciación de las células madre embrionarias, algo esencial para que se las pueda emplear con mayor seguridad en la terapia celular.